2. 中国科学院武汉病毒研究所 病毒学国家重点实验室 武汉 430071
2. State Key Laboratory of Virology, Wuhan Institute of Virology, Chinese Academy of Sciences, Wuhan 430071, China
活细胞及活体内生理和病理过程中关键分子事件的实时、可视化探测对理解基本生物学过程、阐释疾病发生机制等具有重要意义。临床上,疾病的诊断及精准治疗也越发依赖影像学手段。分子生物传感器是一种通过产生可视、可定量信号来指示生物物质或生物事件发生及存在的生物分子系统,在活细胞或者活体内能够实时动态示踪生物分子之间的相互作用、生物大分子的定位和运动、生理环境的变化以及一些重要生化反应等。近20年的研究充分表明分子生物传感器是分子事件“眼见为实”的强有力工具。本文将聚焦其中研究热度较高的分子信标(molecular beacons,MB)、共振能量转移系统(包括荧光共振能量转移和生物发光共振能量转移)和分子荧光互补系统(如双分子荧光互补、三分子荧光互补等),重点阐述这几种分子生物传感器的原理、特点、活细胞中的应用,探讨可能面临的挑战及未来发展方向等。
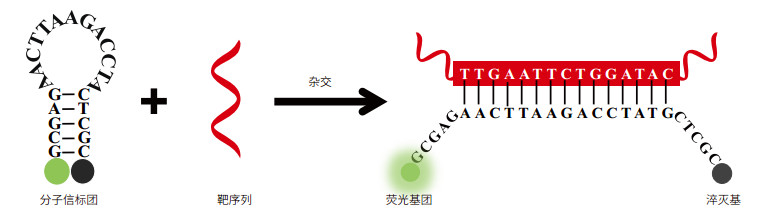
1 分子信标 1.1 分子信标原理分子信标是由Tyagi和Kramer[1]于1996年根据荧光共振能量转移原理首次设计的一种具有茎环结构的寡核苷酸荧光探针,亦称发夹探针。经典的分子信标通常为25-35个核苷酸,包括环形区(Loop)、茎干区(Stem)及其所连接的荧光分子和淬灭分子(图 1)。其中,环形区由15-25个核苷酸组成,可特异性结合靶分子序列。茎干区是5-7个互补碱基对,可在分子信标与靶分子结合过程中发生可逆性解离,用于维持分子特殊形状。荧光基团和荧光淬灭基团则分别位于茎干区的两个末端[2]。当无目标序列存在时,分子信标呈现自由状态,茎干区互补碱基发生结合,促使分子信标形成发夹结构。此时,荧光分子和淬灭分子距离最为接近(约6-10 nm),促发荧光共振能量转移,即:荧光分子发出的荧光被淬灭分子吸收,并以热的形式散发,荧光几乎完全淬灭。相反,当存在目标序列时,分子信标的环形区与靶分子发生杂交,茎干中的互补区打开,由于荧光分子远离淬灭分子,分子信标的荧光近乎100%恢复,其荧光强度与溶液中靶分子浓度呈正相关。
1.2 分子信标应用分子信标具有高灵敏度、高特异性、无须除去冗余等优点。检测聚合酶链式反应(PCR)过程中的扩增产物是分子信标问世的初衷。如今,早非局限于此。研究者通过改造经典的分子信标结构、发展多种荧光分子和淬灭分子、偶联纳米材料(如金纳米粒子/石墨烯、碳纳米管等)等,已成功构建出各种具有优越性能的新型分子信标探针,并将其广泛应用于基因诊断、核酸检测、分子示踪等多个领域。本文主要聚焦分子信标在活细胞内分子影像中的应用。
(1)分子信标在活细胞内示踪mRNA的研究最为丰富。 1998年,Sokol等[3]首次实时示踪了RNA-DNA杂交,在荧光显微镜下检测到原癌基因vav的10个mRNA分子,即0.1 ag(1 ag = 1×10–18 g)的分子信标所产生的杂交信号。随后,Bratu等[4]开辟了利用分子信标在活细胞中研究mRNA定位、转运的先河。该团队在黑腹果蝇卵母细胞中观察了天然mRNA的运动,并发现mRNA 3′端非翻译区的遗传操作或细胞内微管蛋白网络的化学扰动可以明显改变mRNA的分布。Vargas等[5]则通过分子信标追踪活细胞中mRNA分子,进一步对mRNA-蛋白复合物从转录位点到核孔的转运行为进行了可视化研究,提示其转运过程依赖于酶促反应。笔者团队利用分子信标技术率先建立了一种在活的寄主细胞内病毒核酸可视化的方法,在荧光显微镜下可直观地看到脊髓灰质炎病毒的正链RNA和甲型流感病毒mRNA的动态行为,同时结合其他实验手段,探讨了病毒核酸转运机制[6, 7]。
(2)分子信标还较为普遍地用于细胞内蛋白质、ATP等分子的检测与示踪。这主要通过分子信标与适配子形成的适配子信标(aptamer beacon)来完成。Yamamoto等[8]首次证明了将目标物的适配子结合位点插入分子信标中的可能性,构建了靶向HIV病毒Tat蛋白的高亲和力适配子信标。与传统的分子信标相比,这些信标与目标物序列的匹配数目减少了一半(8个碱基)。笔者团队利用相同策略,构建了特异识别HIV逆转录酶的适配体分子信标,进而实现了活细胞中HIV逆转录酶的可视化[9]。Zhang等[10]构建了ATP的分子信标检测系统,实现了在白血病细胞K562和乳腺癌细胞4T1中ATP的检测,体外检测下线低至(1.1-3.2)×10-7 mol/L。Tan等[11]和Liu等[12]分别将氧化石墨烯作为ATP分子信标的淬灭分子,获得了更加稳定、特异的适配子信标,前者可以半定量检测活细胞内ATP,后者可响应5 mmol/L Ca2+诱导的ATP升高。
目前,针对性发展多功能分子信标,在单细胞、单分子水平示踪关键分子事件的研究工作已经萌芽。例如,Li等[13]设计了一种双功能分子信标,能够同时检测并抑制斑马鱼中的microRNA。Chen等[14]利用分子信标最小工程化①地点亮单分子RNA,通过成像技术深入研究了单细胞非编码RNA(lncRNA)的动力学和定位。这些方向可能成为分子信标在细胞成像领域应用中的主流。
① 即只需要极少的分子工程改造
2 共振能量转移系统共振能量转移系统是利用能量转移原理和基因操纵技术构建的分子传感体系,主要包括荧光共振能量转移(fluorescence resonance energy transfer,FRET)和生物发光共振能量转移(bioluminescent resonance energy transfer,BRET)。
2.1 荧光共振能量转移 2.1.1 原理FRET是1948年由Förster[15]提出的一种基于能量转移来测量分子间距离的技术。其基本原理是当供体的荧光发射光谱与受体的吸收光谱重叠,并且供体和受体的距离合适时(一般小于10 nm),就会发生一种非放射性的能量转移。如图 2所示,当以适当频率的激发光照射供体时,供体会产生振荡偶极子并与相邻受体的偶极子产生共振。供体荧光团的能量由于偶极子之间的相互作用非放射性地转移到受体荧光团。总体而言,产生FRET现象需要同时具备两个条件:(1)供体分子具有较高的量子产率,其发射光光谱与受体分子的激发光光谱有效重叠;(2)供体和受体分子都处于被激发状态,两者之间距离在1-10 nm之间[16]。
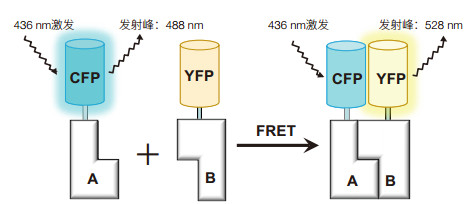
|
| 图 2 FRET技术研究蛋白质之间相互作用 A和B是一对相互作用的蛋白 |
在生物学研究领域,FRET技术是研究生物大分子间相互作用,特别是在活细胞生理条件下研究蛋白质-蛋白质间相互作用的有力工具。FRET供、受体主要是荧光蛋白、有机荧光分子、无机纳米荧光材料等,其发生需要外界光源激发荧光供体。青色荧光蛋白(CFP)和黄色荧光蛋白(YFP)是最常用的一对供受体。由于FRET严格依赖空间距离(1-10 nm),且大多数生物分子介于该尺寸之间,因此,FRET是一把丈量分子空间距离的光学“分子尺”,可以解决生物学中诸多难题,如蛋白质动力学、蛋白质构象变化等。有别于经典的分子发射光谱分析法,FRET最大优点是,该技术利用信号比值而非信号强度进行数据分析,消除了取样误差而造成的信号间差异。近年来,由于新型荧光材料的出现、光学技术的进步以及显微成像工具的发展,FRET技术在空间分辨率、检测灵敏度等方面有了极大的提升,目前已实现单分子FRET(SmFRET)[17]。
2.1.3 应用FRET在活细胞中的应用非常之多,即利用两个蛋白分子之间相互作用的空间信息以回答分子运动、蛋白构象、转录调控等相关生物学问题。技术上,主要通过显微镜,利用受体漂白、导致的供体荧光增强、供体荧光漂白、受体荧光寿命增长、供-受体荧光强度比率法等多种手段来检测FRET现象[18],在此介绍部分代表性研究。
(1)活细胞内生理状态下的离子检测是FRET技术较为经典的应用。例如,Myawaki等[19]发展了一种由CFP、YFP、钙调蛋白和钙调蛋白结合肽M13组成的活细胞钙离子指示剂。其原理是,细胞里的Ca2+促进钙调蛋白与M13结合,进而增加了荧光蛋白之间的荧光共振能量转移,因此可通过检测FRET现象和数据校正,实现细胞器中钙离子的监测与定量。无独有偶,Zhang等[20]利用FRET探针对活细胞中Hg2+进行了高灵敏检测。另外,针对活细胞中ATP、pH值等重要生理参数,均有巧妙的FRET示踪体系被相继报道[21-23]。
(2)FRET无疑是研究蛋白质互作的最好手段之一。蛋白质-蛋白质相互作用涉及生命体中众多生理过程和信号转导过程,在细胞活动中扮演关键性角色。利用FRET技术检测蛋白质互作的策略通常是利用基因操纵,将两个目标蛋白分别偶联一个供体或受体,并在细胞内融合表达。在特定时空及生理条件下,如果存在互作,供体和受体会相互靠近产生FRET现象,反之则无。利用这一策略,细胞中多种蛋白之间的互作已被鉴定和示踪。例如,高迁移率族蛋白1(HMGB1)、Toll样受体(TLR)与晚期糖基化终产物受体(RAGE)之间的相互作用[24];亚细胞结构中表皮生长因子受体(EGFR)与和其结合蛋白Grb2之间的互作[25];基质相互作用分子1(STIM1)之间的寡聚化及其易位、靶向等[26]。
除此之外,FRET也普遍用于活细胞中RNA检测、蛋白质动力学等相关研究[27-29]。
2.2 生物发光共振能量转移BRET于1999年问世,与FRET基本原理相似,有异曲同工之妙[30]。BRET体系中的供体主要是荧光素酶(Luc)。其中,海肾荧光素酶(Rluc)以腔肠素(Coelenterazine)为底物,萤火虫荧光素酶(Fluc)以D-Luciferin和ATP为底物,海萤荧光素酶(Cluc)则以海萤荧光素为底物,这些均是BRET常用供体。如图 3所示,这些底物及其衍生物会被相应的荧光素酶催化并氧化发光。由于A-B分子的相互作用,供受体相互靠近。当两者距离1-10 nm时,BRET现象产生,受体发光。底物饱和状态下,受体荧光强度与酶浓度呈正相关。较FRET比,由于BRET无须外界光源激发供体,因此可避免强光源导致的光漂白、光吸收、组织损伤等现象,同时能够降低因生物体内自发荧光产生的背景信号[31]。
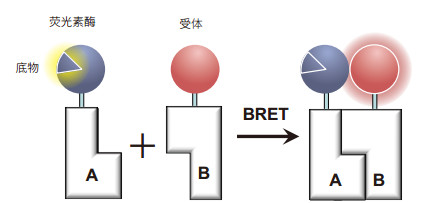
|
| 图 3 BRET工作原理图 与A蛋白融合表达的荧光素酶催化其底物氧化发光,经A-B蛋白之间相互作用后,供体荧光素酶与受体相互靠近,发生共振能量转移,受体产生荧光 |
在活细胞影像的应用方面,BRET和FRET大致相同,如蛋白质相互作用、ATP检测、蛋白质活性测定等[32-35]。值得一提的是,在富含血细胞或血红色素的组织及活体成像中,由于生物体内光吸收、自发荧光的减少,使BRET在信噪比方面具有明显优势[36]。
3 分子互补系统分子互补系统是利用蛋白质片段互补技术构建的分子传感体系。本文着重阐述的是以荧光蛋白为信号元件的分子互补系统,包括双分子荧光互补(bimolecular fluorescence complementation, BiFC)、三分子荧光互补(trimolecular fluorescence complementation,TriFC)等。
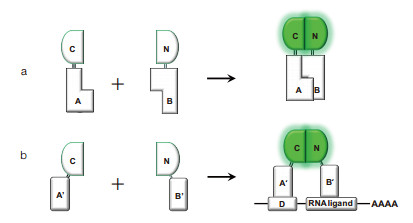
3.1 双分子荧光互补 3.1.1 原理BiFC起源于蛋白质片段互补技术,由Ghosh等[37]于2000年首次报道。如图 4a所示[38],荧光蛋白在特定区域(通常是loop区)通过基因剪接形成两个非荧光片段,称为N片段和C片段。N、C两个片段通过Linker肽分别与一对相互作用的靶蛋白融合表达。由于靶蛋白对之间的相互作用,荧光蛋白N片段和C片段会紧密靠近,形成空间互补,进而发生分子重建恢复构象,并在合适的激发波长下产生荧光。反之,若靶蛋白之间不存在相互作用,则无荧光产生。
BiFC可应用于体内和体外,具有高灵敏、低噪声等特点。BiFC分子元件预先通过基因操纵导入细胞中表达,无须外源添加即可直接利用荧光观测活细胞或活体中生物分子的相互作用及其空间位置信息,因此避免了外源物质对细胞的干扰和观察的失真。与FRET和BRET技术相比,BiFC不用复杂的数据处理,常规荧光显微镜即可满足。利用不同颜色的荧光互补系统,还可以在活细胞内同时检测多个蛋白的相互作用。鉴于这些特点,BiFC被广泛用于生物分子相互作用的研究,特别是细胞内之间、细胞与环境物质之间的相互作用。
目前所报道的BiFC系统通常不可逆、即结合重构后很难再被打开。这个特点使得BiFC灵敏度很高,可检测KD接近1 mmol/L的弱相互作用,但也阻碍了BiFC监测蛋白质相互作用的动态变化。为此,Tchekanda等[39]利用基因改造的近红外荧光蛋白突变单体IFP1.4发展了可逆的BiFC系统,进一步扩展了BiFC技术的应用。
3.1.3 应用BiFC常用于研究蛋白质寡聚化(protein oligomerization)。其中,α-突触核蛋白(α-synuclein)的错误折叠--寡聚化和纤维化被认为是帕金森和其他相关神经疾病发生和发展的核心事件。Outeiro等[40]通过BiFC直观地观察了突触核蛋白在活细胞中的寡聚化,并发现蛋白寡聚化导致的细胞毒性增加可通过Hsp70来缓解。除此之外,G蛋白偶联受体(GPCRs)的寡聚化是又一重要研究领域。GPCRs及相关信号转导涉及诸多疾病,其寡聚化在调节受体药理学和功能中具有重要作用。目前大约30%-40%药物的细胞靶标针对GPCRs而设计[41, 42]。在该领域,有大量基于BiFC的研究与发现,包括GPCR二聚化[43]和二聚化的抑制[44]、腺苷A2A受体的分布和其在质膜上的组装[45, 46]、利用GPCR二聚体成像研究神经肽Y Y1/Y5受体异二聚体的药理学等[47]。Cirulela和Vilardaga[48]更是详细总结了BiFC在GPCR研究中的应用。因此,BiFC不仅是示踪蛋白寡聚化的重要工具,更是寻找相关疾病治疗靶标和药物的有效手段。
BiFC也广泛用于研究病毒与宿主之间的相互作用。这些研究对于理解病毒的生命周期和致病机制具有重要意义。例如,Hemerka等[49]利用BiFC揭示了流感病毒聚合酶复合物PA与PB2亚基之间前所未知的相互作用,并对PA-PB2二元复合物的亚细胞定位与进核进行了详细分析,其研究思路为进一步探索PA-PB2互作与甲型流感病毒聚合酶活性、病毒复制的生物相关性提供了一个框架。目前,利用BiFC对人类病毒的研究还涉及EpsteinBarr病毒潜伏膜蛋白1的表征[50]、基于Beclin1-Bcl2复合物解离的自噬抑制剂药物筛选[51]、HIV病毒非结构性蛋白Vpr分子之间的互作等[52]。
3.2 分子互补系统的发展 3.2.1 远红外和近红外BiFC在哺乳动物中存在细胞成像的近红外(NIR)光学窗口(650-900 nm),以避免生物体内水、血红蛋白等分子对光的吸收[53, 54]。为了解决这个问题,笔者团队利用mCherry(λex/λem = 587 nm/610 nm)、mNeptune(λex/λem = 600 nm/650 nm)等荧光蛋白开发了系列长波长荧光分子互补系统[55, 56]。另外,为了更好地实现深部组织成像,我们打破利用GFP-like荧光蛋白构建BiFC这一常规,创新性的发展了以细菌光敏色素iRFP为基础的近红外BiFC系统(λex/λem = 690 nm/713 nm)。该工作为生理条件下研究蛋白质相互作用提供了强有力工具,同时也为活细胞中的药物评价提供了新策略[57]。
3.2.2 三分子互补系统鉴于细胞内未知的蛋白质-RNA之间互作,基于BiFC建立的TriFC系统应运而生。如图 4b所示,在TriFC系统中,荧光蛋白的C片段通过已知蛋白质(A ′)和RNA(D)之间的相互作用,附着于报告mRNA。N片段与RNA -结合蛋白B′融合表达。如果RNA结合蛋白与报告基因mRNA中的靶序列相互作用,则N和C两片段紧密接近,恢复荧光蛋白构象产生荧光[58]。TriFC不仅可用于鉴定RNA-蛋白质相互作用,还可分析活细胞中RNA-蛋白质相互作用的定位和动力学。大量研究已验证该方法的实用性[59-61]。其中,笔者团队利用TriFC重点研究了流感病毒和HIV病毒mRNA与宿主蛋白之间的相互作用,并阐释了相关生物学机制[55, 62]。
最近,笔者团队还发展了三片段荧光互补系统(three-fragment fluorescence complementation,TFFC),用于蛋白三聚体的成像研究。我们把具有光转换和光激活特性的荧光蛋白mIrisFP拆分为3个片段,结合超分辨成像,在单分子水平示踪了活细胞中G蛋白三聚体亚基间的相互作用[63]。除此之外,在荧光蛋白第10个和第11个β-strand之间“切割形成的分子互补系统”(split fluorescent proteins complementation)也被用于活细胞可视化研究,如蛋白质的定位、胞浆肽的输送等[64-66]。
4 结语与展望近20年来,上述以荧光为信号的分子生物传感技术进展迅速,大量相关研究论文被发表,涵盖了材料、化学、物理、生物医学、生命科学等多个学科。从活细胞层面的应用态势来看,利用多色标记,结合超分辨成像,在单细胞、单分子水平实时动态观察多个关键分子事件将成为主流。但是,这些分子生物传感器在组织、器官和活体的深层次应用仍然面临严峻挑战。提高信噪比、避免生物物质光吸收、允许长时示踪都还是难题。亟待创造生物相容性好、亮度高、抗淬灭荧光探针,提出超高灵敏传感新体系,改进成像数据算法,也需要进一步推动光学技术、成像工具的发展等。随着技术进步,分子生物传感器将使我们前所未有地实时观察获取生命过程深层次信息,从而回答一系列重要的生命科学基本问题,并将拓展到临床应用,为人类健康服务。
| [1] |
Tyagi S, Kramer F R. Molecular beacons:probes that fluoresce upon hybridization[J]. Nature Biotechnology, 1996, 14: 303-308. DOI:10.1038/nbt0396-303 |
| [2] |
Tyagi S, Kramer F R. Molecular beacons in diagnostics[J]. F1000 Medicine Reports, 2012, 4: 10. |
| [3] |
Sokol D L, Zhang X, Lu P, et al. Real time detection of DNA·RNA hybridization in living cells[J]. PNAS, 1998, 95: 11538-11543. DOI:10.1073/pnas.95.20.11538 |
| [4] |
Bratu D P, Cha B J, Mhlanga M M, et al. Visualizing the distribution and transport of mRNAs in living cells[J]. PNAS, 2003, 100: 13308-13313. DOI:10.1073/pnas.2233244100 |
| [5] |
Vargas D Y, Raj A, Marras S A E, et al. Mechanism of mRNA transport in the nucleus[J]. PNAS, 2005, 102: 17008-17013. DOI:10.1073/pnas.0505580102 |
| [6] |
Wang W, Cui Z Q, Han H, et al. Imaging and characterizing influenza A virus mRNA transport in living cells[J]. Nucleic Acids Research, 2008, 36: 4913-4928. DOI:10.1093/nar/gkn475 |
| [7] |
Cui Z Q, Zhang Z P, Zhang X E, et al. Visualizing the dynamic behavior of poliovirus plus-strand RNA in living host cells[J]. Nucleic Acids Research, 2005, 33: 3245-3252. DOI:10.1093/nar/gki629 |
| [8] |
Yamamoto R, Baba T, Kumar P K R. Molecular beacon aptamer fluoresces in the presence of Tat protein of HIV-1[J]. Genes to Cells, 2010, 5: 389-396. |
| [9] |
Liang Y, Zhang Z P, Wei H P, et al. Aptamer beacons for visualization of endogenous protein HIV-1 reverse transcriptase in living cells[J]. Biosensors & Bioelectronics, 2011, 28: 270-276. |
| [10] |
Zhang S S, Yan Y M, Bi S. Design of molecular beacons as signaling probes for adenosine triphosphate detection in cancer cells based on chemiluminescence resonance energy transfer[J]. Analytical Chemistry, 2009, 81: 8695-8701. DOI:10.1021/ac901759g |
| [11] |
Tan X H, Chen T, Xiong X L, et al. Semiquantification of ATP in live cells using nonspecific desorption of DNA from graphene oxide as the internal reference[J]. Analytical Chemistry, 2012, 84: 8622-8627. DOI:10.1021/ac301657f |
| [12] |
Liu Z B, Chen S S, Liu B W, et al. Intracellular detection of ATP using an aptamer beacon covalently linked to graphene oxide resisting nonspecific probe displacement[J]. Analytical Chemistry, 2014, 86: 12229-12235. DOI:10.1021/ac503358m |
| [13] |
Li W M, Chan C M, Miller A L, et al. Dual functional roles of molecular beacon as a MicroRNA detector and inhibitor[J]. Journal of Biological Chemistry, 2017, 292: 3568-3580. DOI:10.1074/jbc.M116.765776 |
| [14] |
Chen M M, Ma Z, Wu X T, et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level[J]. Scientific Reports, 2017, 7: 1550. DOI:10.1038/s41598-017-01740-1 |
| [15] |
Förster T. Zwischenmolekulare Energiewanderung und Fluoreszenz[J]. Annals in Physics, 1948, 2: 55-75. |
| [16] |
Wu P G, Brand L. Resonance energy transfer:methods and applications[J]. Analytical Biochemistry, 1994, 218: 1-13. DOI:10.1006/abio.1994.1134 |
| [17] |
Jares-Erijman E A, Jovin T M. FRET imaging[J]. Nature Biotechnology, 2003, 21: 1387-1395. DOI:10.1038/nbt896 |
| [18] |
Wouters F S, Verveer P J, Bastiaens P I H. Imaging biochemistry inside cells[J]. Trends in Cell Biology, 2001, 11: 203-211. DOI:10.1016/S0962-8924(01)01982-1 |
| [19] |
Miyawaki A, Llopis J, Heim R, et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin[J]. Nature, 1997, 388: 882-887. DOI:10.1038/42264 |
| [20] |
Zhang X L, Xiao Y, Qian X H. A Ratiometric fluorescent probe based on FRET for imaging Hg2+ ions in living cells[J]. Angewandte Chemie International Edition, 2008, 47: 8025-8029. DOI:10.1002/anie.v47:42 |
| [21] |
Modi S, Swetha M G, Goswami D, et al. A DNA nanomachine that maps spatial and temporal pH changes inside living cells[J]. Nature Nanotechnology, 2009, 4: 325-330. DOI:10.1038/nnano.2009.83 |
| [22] |
Imamura H, Nhat K P H, Togawa H, et al. Visualization of ATP levels inside single living cells with fluorescence resonance energy transfer-based genetically encoded indicators[J]. PNAS, 2009, 106: 15651-15656. DOI:10.1073/pnas.0904764106 |
| [23] |
Dennis A M, Rhee W J, Sotto D, et al. Quantum dot-fluorescent protein FRET probes for sensing intracellular pH[J]. ACS Nano, 2012, 6: 2917-2924. DOI:10.1021/nn2038077 |
| [24] |
Park J S, Gamboni-Robertson F, He Q B, et al. High mobility group box 1 protein interacts with multiple Toll-like receptors[J]. American Journal of Physiology-Cell Physiology, 2006, 290: C917-C924. |
| [25] |
Sorkin A, McClure M, Huang F T, et al. Interaction of EGF receptor and Grb2 in living cells visualized by fluorescence resonance energy transfer (FRET) microscopy[J]. Current Biology, 2000, 10: 1395-1398. DOI:10.1016/S0960-9822(00)00785-5 |
| [26] |
Liou J, Fivaz M, Inoue T, et al. Live-cell imaging reveals sequential oligomerization and local plasma membrane targeting of stromal interaction molecule 1 after Ca2+ store depletion[J]. PNAS, 2007, 104: 9301-9306. DOI:10.1073/pnas.0702866104 |
| [27] |
Santangelo P J, Nix B, Tsourkas A, et al. Dual FRET molecular beacons for mRNA detection in living cells[J]. Nucleic Acids Research, 2004, 32. |
| [28] |
He L, Lu D Q, Liang H, et al. Fluorescence resonance energy transfer-based DNA tetrahedron nanotweezer for highly reliable detection of tumor-related mRNA in living cells[J]. ACS Nano, 2017, 11: 4060-4066. DOI:10.1021/acsnano.7b00725 |
| [29] |
Ma Y Q, Pandzic E, Nicovich P R, et al. An intermolecular FRET sensor detects the dynamics of T cell receptor clustering[J]. Nature Communications, 2017, 8: 15100. DOI:10.1038/ncomms15100 |
| [30] |
Xu Y, Piston D W, Johnson C H. A bioluminescence resonance energy transfer (BRET) system:Application to interacting circadian clock proteins[J]. PNAS, 1999, 96: 151-156. DOI:10.1073/pnas.96.1.151 |
| [31] |
Scott D, Dikici E, Ensor M, et al. Bioluminescence and its impact on bioanalysis[J]. Annual Review of Analytical Chemistry, 2011, 4: 297-319. DOI:10.1146/annurev-anchem-061010-113855 |
| [32] |
Pfleger K D G, Seeber R M, Eidne K A. Bioluminescence resonance energy transfer (BRET) for the real-time detection of protein-protein interactions[J]. Nature Protocols, 2006, 1: 337-345. DOI:10.1038/nprot.2006.52 |
| [33] |
Xia Z Y, Rao J H. Biosensing and imaging based on bioluminescence resonance energy transfer[J]. Current Opinion in Biotechnology, 2009, 20: 37-44. DOI:10.1016/j.copbio.2009.01.001 |
| [34] |
den Hamer A, Dierickx P, Arts R, et al. Bright bioluminescent BRET sensor proteins for measuring intracellular caspase activity[J]. ACS Sensors, 2017, 2: 729-734. DOI:10.1021/acssensors.7b00239 |
| [35] |
Yoshida T, Kakizuka A, Imamura H. BTeam, a novel BRET-based biosensor for the accurate quantification of ATP concentration within living cells[J]. Scientific Reports, 2016, 6: 39618. DOI:10.1038/srep39618 |
| [36] |
Dragulescu-Andrasi A, Chan C T, De A, et al. Bioluminescence resonance energy transfer (BRET) imaging of protein-protein interactions within deep tissues of living subjects[J]. PNAS, 2011, 108: 12060-12065. DOI:10.1073/pnas.1100923108 |
| [37] |
Ghosh I, And A D H, Regan L. Antiparallel leucine zipper-directed protein reassembly:application to the green fluorescent protein[J]. Journal of the American Chemical Society, 2000, 122: 5658-5659. DOI:10.1021/ja994421w |
| [38] |
Zhang X E, Cui Z, Wang D. Sensing of biomolecular interactions using fluorescence complementing systems in living cells[J]. Biosens Bioelectron, 2016, 76: 243-250. DOI:10.1016/j.bios.2015.07.069 |
| [39] |
Tchekanda E, Sivanesan D, Michnick S W. An infrared reporter to detect spatiotemporal dynamics of protein-protein interactions[J]. Nature Methods, 2014, 11: 641. DOI:10.1038/nmeth.2934 |
| [40] |
Outeiro T F, Putcha P, Tetzlaff J E, et al. Correction:formation of toxic oligomeric α-synuclein species in living cells[J]. PLoS One, 2008, 3: e1867. DOI:10.1371/journal.pone.0001867 |
| [41] |
Bosch D E, Siderovski D P. G protein signaling in the parasite Entamoeba histolytica[J]. Experimental & Molecular Medicine, 2013, 45: e15. |
| [42] |
Overington J P, Al-Lazikani B, Hopkins A L. How many drug targets are there?[J]. Nature Reviews Drug Discovery, 5: 993-996. DOI:10.1038/nrd2199 |
| [43] |
Kosel D, Heiker J T, Juhl C, et al. Dimerization of adiponectin receptor1 is inhibited by adiponectin[J]. Journal of Cell Science, 2010, 123: 1320-1328. DOI:10.1242/jcs.057919 |
| [44] |
Ejendal K F, Conley J M, Hu C D, et al. Bimolecular fluorescence complementation analysis of G protein-coupled receptor dimerization in living cells[J]. Methods in Enzymology, 2013, 521: 259-279. DOI:10.1016/B978-0-12-391862-8.00014-4 |
| [45] |
Gandia J, Galino J, Amaral O B, et al. Detection of higher-order G protein-coupled receptor oligomers by a combined BRET-BiFC technique[J]. FEBS Letters, 2008, 582: 2979-2984. DOI:10.1016/j.febslet.2008.07.045 |
| [46] |
Vidi P A, Chemel B R, Hu C D, et al. Ligand-dependent oligomerization of dopamine D(2) and adenosine A(2A) receptors in living neuronal cells[J]. Molecular Pharmacology, 2008, 74: 544. DOI:10.1124/mol.108.047472 |
| [47] |
Kilpatrick L E, Humphrys L J, Holliday N D. A G protein coupled receptor dimer imaging assay reveals selectively modified pharmacology of neuropeptide Y Y1/Y5 receptor heterodimers[J]. Molecular Pharmacology, 2015, 87: 718-732. DOI:10.1124/mol.114.095356 |
| [48] |
Ciruela F, Vilardaga J P. Lighting up multiprotein complexes:lessons from GPCR oligomerization[J]. Trends in Biotechnology, 2010, 28: 407-415. DOI:10.1016/j.tibtech.2010.05.002 |
| [49] |
Hemerka J N, Wang D, Weng Y, et al. Detection and characterization of influenza A virus PA[J]. Journal of Virology, 2009, 83: 3944. DOI:10.1128/JVI.02300-08 |
| [50] |
Talaty P, Emery A, Everly D N. Characterization of the latent membrane protein 1 signaling complex of Epstein-Barr virus in the membrane of mammalian cells with bimolecular fluorescence complementation[J]. Virology Journal, 2011, 8: 1-15. DOI:10.1186/1743-422X-8-1 |
| [51] |
Dai J P, Zhao X F, Zeng J, et al. Drug screening for autophagy inhibitors based on the dissociation of Beclin1-Bcl2 complex using BiFC technique and mechanism of eugenol on anti-influenza A virus activity[J]. PLoS One, 2013, 8: e61026. DOI:10.1371/journal.pone.0061026 |
| [52] |
Venkatachari N J, Walker L A, Tastan O, et al. Human immunodeficiency virus type 1 Vpr:oligomerization is an essential feature for its incorporation into virus particles[J]. Virology Journal, 2010, 7: 1-11. DOI:10.1186/1743-422X-7-1 |
| [53] |
Weissleder R. A clearer vision for in vivo imaging[J]. Nature Biotechnology, 2001, 19: 316. DOI:10.1038/86684 |
| [54] |
Weissleder R, Ntziachristos V. Shedding light onto live molecular targets[J]. Nature Medicine, 2003, 9: 123-128. DOI:10.1038/nm0103-123 |
| [55] |
Han Y, Wang S, Zhang Z, et al. In vivo imaging of protein-protein and RNA-protein interactions using novel far-red fluorescence complementation systems[J]. Nucleic Acids Research, 2014, 42: e103. DOI:10.1093/nar/gku408 |
| [56] |
Fan J Y, Cui Z Q, Wei H P, et al. Split mCherry as a new red bimolecular fluorescence complementation system for visualizing protein-protein interactions in living cells[J]. Biochemical and Biophysical Research Communications, 2008, 367: 47-53. DOI:10.1016/j.bbrc.2007.12.101 |
| [57] |
Chen M, Li W, Zhang Z, et al. Novel near-infrared BiFC systems from a bacterial phytochrome for imaging protein interactions and drug evaluation under physiological conditions[J]. Biomaterials, 2015, 48: 97. DOI:10.1016/j.biomaterials.2015.01.038 |
| [58] |
Rackham O, Brown C M. Visualization of RNA-protein interactions in living cells:FMRP and IMP1 interact on mRNAs[J]. The EMBO Journal, 2004, 23: 3346-3355. DOI:10.1038/sj.emboj.7600341 |
| [59] |
Valencia-Burton M, McCullough R M, Cantor C R, et al. RNA visualization in live bacterial cells using fluorescent protein complementation[J]. Nature Methods, 2007, 4: 421-427. |
| [60] |
Milev M P, Brown C M, Mouland A J. Live cell visualization of the interactions between HIV-1 Gag and the cellular RNA-binding protein Staufen1[J]. Retrovirology, 2010, 7. |
| [61] |
Ortega A D, Willers I M, Sala S, Cuezva J M. Human G3BP1 interacts with b-F1-ATPase mRNA and inhibits its translation[J]. Journal of Cell Science, 2010, 123: 2685-2696. DOI:10.1242/jcs.065920 |
| [62] |
Yin J, Zhu D H, Zhang Z P, et al. Imaging of mRNA-protein interactions in live cells using novel mCherry trimolecular fluorescence complementation systems[J]. PLoS One, 2013, 8(11): e80851. DOI:10.1371/journal.pone.0080851 |
| [63] |
Chen M, Liu S, Li W, et al. Three-fragment fluorescence complementation coupled with photoactivated localization microscopy for nanoscale imaging of ternary complexes[J]. ACS Nano, 2016, 10: 8482-8490. DOI:10.1021/acsnano.6b03543 |
| [64] |
Schmidt S, Adjobo-Hermans M J W, Wallbrecher R, et al. Detecting cytosolic peptide delivery with the GFP complementation assay in the low micromolar range[J]. Angewandte Chemie International Edition, 2015, 54: 15105-15108. DOI:10.1002/anie.201505913 |
| [65] |
Kamiyama D, Sekine S, Barsi-Rhyne B, et al. Versatile protein tagging in cells with split fluorescent protein[J]. Nature Communications, 2016, 7: 11046. DOI:10.1038/ncomms11046 |
| [66] |
Feng S Y, Sekine S, Pessino V, et al. Improved split fluorescent proteins for endogenous protein labeling[J]. Nature Communications, 2017, 8: 370. DOI:10.1038/s41467-017-00494-8 |